 Situer lanalyse phylogénétique entre les sciences historiques et
Situer lanalyse phylogénétique entre les sciences historiques et
24 mai 2019 tion puis (4) l'interprétation d'un arbre phylogénétique. Nous montrons que l'analyse phylogénétique n'est pas une expérimentation et donc ...
 Méthode du maximum de vraisemblance
Méthode du maximum de vraisemblance
Les arbres phylogénétiques construits avec une méthode quelconque ne sont qu'une estimation de l'histoire évolutive des séquences. Il est donc important de
 cours de phylogénie moléculaire
cours de phylogénie moléculaire
Les nœuds internes sont des unités inexistantes en fait mais des ancêtres hypothétiques qui permettent l'interprétation de l'arbre. Page 10. 10. Dans cet arbre
 Savoir lire un arbre phylogénétique
Savoir lire un arbre phylogénétique
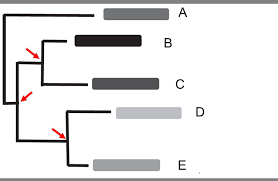
L'arbre phylogénétique présente les relations de parenté entre organismes vivants. Il montre qui est proche de qui et non pas qui descend de qui.
 Recherche automatisée de motifs dans les arbres phylogénétiques
Recherche automatisée de motifs dans les arbres phylogénétiques
24 juil. 2014 Cette interprétation de l'arbre des espèces ne consiste pas seulement à copier en mémoire l'arbre Newick sous forme d'objet Bio++. Ce ...
 Analyse phylogénétique du SARS-CoV-2 en Ontario (Interface
Analyse phylogénétique du SARS-CoV-2 en Ontario (Interface
24 mars 2021 et peut être vue sur l'arbre phylogénétique. L'utilisateur est en ... d'interprétation de la part des utilisateurs. Toutefois il est ...
 Présentation
Présentation
À partir de cette matrice nous allons construire l'arbre phylogénétique. Les 5 animaux vont être d'abord rassemblés dans un même groupe (ficelle bleue)
 Introduction aux méthodes pour la phylogénie moléculaire
Introduction aux méthodes pour la phylogénie moléculaire
Strict consensus of six trees based on equal weights parsimony analysis of the entire data set of three exons and two introns. • Tout arbre phylogénétique ...
 Tit Initiation à lalignement de séquence et à la phylogénie
Tit Initiation à lalignement de séquence et à la phylogénie
1 mai 2010 Un arbre décrivant les relations évolutives entre les séquences. i.e. un arbre phylogénétique. ... Interprétation. Visualisation. Reconstruction d ...
 Savoir lire un arbre phylogénétique
Savoir lire un arbre phylogénétique
L'arbre phylogénétique présente les relations de parenté entre organismes vivants. Il montre qui est proche de qui et non pas qui descend de qui.
 Comprendre et Exploiter un Arbre Phylogénétique
Comprendre et Exploiter un Arbre Phylogénétique
On représente aujourd'hui les liens de parenté entre les espèces sous la forme d'arbres phylogénétiques. Le mot phylogénétique vient de la phylogénie (du grec.
 Introduction à la bioinformatique Construction des arbres
Introduction à la bioinformatique Construction des arbres
Les arbres non racinés ne sont pas réellement des arbres phylogénétiques car Comment enraciner un arbre phylogénétique ? ... Interprétation du Bootstrap ...
 cours de phylogénie moléculaire
cours de phylogénie moléculaire
initiales va permettre d'aboutir à des interprétations et des concluions passer à la construction de l'arbre phylogénétique et de bien régler les ...
 Situer lanalyse phylogénétique entre les sciences historiques et
Situer lanalyse phylogénétique entre les sciences historiques et
24 mai 2019 tion puis (4) l'interprétation d'un arbre phylogénétique. ... Phylogenetic analysis is one of the main methods of evolutionary biology.
 Comment et pourquoi représenter larbre phylogénétique du vivant
Comment et pourquoi représenter larbre phylogénétique du vivant
6 avr. 2015 Les 3 branches du vivant. (animaux protistes et végétaux selon l'interprétation de la fin du XIXème siècle) sont traitées "à égalité". Chez les.
 Construire-et-étudier-un-arbre-phylogénétique.pdf
Construire-et-étudier-un-arbre-phylogénétique.pdf
Un arbre phylogénétique est une représentation graphique de la phylogenèse d'un groupe de taxons. Les sommets représentent les taxons ou les unités
 Phylogénie moléculaire
Phylogénie moléculaire
Les arbres phylogénétiques s'avèrent être des outils cen- puient sur l'interprétation dite subjectiviste des probabili- tés. Nous avons vu plus haut ...
 Introduction à la Phylogénie Moléculaire : Concepts méthodes et
Introduction à la Phylogénie Moléculaire : Concepts méthodes et
Concepts méthodes et interprétation. Du 13 au 18 Decembre 2021. Enseignants : De l'échantillon à la construction d'arbres phylogénétiques.
 Les méthodes probabilistes en phylogénie moléculaire: (1) Les
Les méthodes probabilistes en phylogénie moléculaire: (1) Les
30 nov. 2007 Phylogenetic analysis : models and estimation proce- dures. Evolution 21 : 550-570. FELSENSTEIN J.
 APP3 : Arbres phylogénétiques
APP3 : Arbres phylogénétiques
Fiche méthodologique : Savoir lire un arbre phylogénétique auteur : N Lepouder L'arbre phylogénétique présente les relations de parenté entre organismes vivants Il montre qui est proche de qui et non pas qui descend de qui L'arbre phylogénétique montre l’ordre d’apparition des différents groupes
 APP3 : Arbres phylogénétiques - GitLab
APP3 : Arbres phylogénétiques - GitLab
Un arbre phylogénétique est un arbre schématique qui montre les relations de parenté entre des groupes d’êtres vivants Au sein d’un arbre phylogénétique un groupe d’organismes est dé?ni par une caractéristique commune à tous les organismes de ce groupe Cette caractéristique issue de
 Introduction à la Phylogénie
Introduction à la Phylogénie
T: arbre raciné Soit M un groupe d’espèces (actuelles et ancestrales) M Groupe Monophylétique si le LCA e de M ainsi que tous ses descendants sont dans M Autrement dit M détermine un sous-arbre de T Exemple dans l’arbre des tétrapodes: Mammifères M Groupe Paraphylétique si le LCA e de M est dans M mais que M
 Introduction à la phylogénie*
Introduction à la phylogénie*
La construction d’un arbre par UPGMA sous-entend un modèle d’évolution faisant intervenir l’hypothèse de l’horloge moléculaire: taux de mutation constant UPGMA trouve LE bon arbre ssi il existe un arbre ultramétrique pour D Dé?nition: Soit D une matrice symétrique n X n Un arbre ultramétrique associé à D est un arbre A tel que:
 leay:block;margin-top:24px;margin-bottom:2px; class=tit bzhsvte-monsitecomFiche méthodologie de construction d’un arbre phylogénétique
leay:block;margin-top:24px;margin-bottom:2px; class=tit bzhsvte-monsitecomFiche méthodologie de construction d’un arbre phylogénétique
Fiche méthodologie de constuction d’un abe phylogénétiue (3ème) Nomenclature : Ab e phylogénétiue = abe de paenté = abe d’évolution = cladogramme Un arbre phylogénétique permet de représenter simplement des liens hypothétiques entre des espèces Pour
Comment définir un arbre phylogénétique?
- Au sein d’un arbre phylogénétique, un groupe d’organismes est dé?ni par unecaractéristique, commune à tous les organismes de ce groupe. Cette caractéristique, issue de l’évolution, peut être morphologique (par exemple le « pouce opposable » pour le groupe des primates), ou génétique (présence de certains gènes ou protéines).
Qu'est-ce que l'arbre phylogénétique?
- Unarbre phylogénétiqueest un arbre schématique qui montre les relations de parenté entre des groupes d’êtres vivants. Au sein d’un arbre phylogénétique, un groupe d’organismes est dé?ni par unecaractéristique, commune à tous les organismes de ce groupe.
Comment augmenter le temps de calcul de la phylogénie ?
- L’exploration de chaque position de chaque séquence pour la détermination éventuelle d’une insertion augmente d’un facteur 2N le temps de calcul. La programmation dynamique est un moyen de limiter cette augmentation pour conserver un temps de calcul de l’ordre de N². Introduction générale à la phylogénie. Acquisition du jeu de données.
Comment savoir si une matrice possède un arbre phylogénétique?
- Cette matrice possède un arbre phylogénétique si 1) T a n feuilles correspondant à chacune des espèces 2) Chaque caractère ou trait est l’étiquette d’une arête 3) Les étiquettes de la racine à la feuille i énumèrent tous les caractères présents dans l’espèce i IFT6291, A2006, Sylvie Hamel Université de Montréal
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 1 ComprendreetExploiterunArbrePhylogénétique Menu 1. L'arbre phylogénétique : un document qui raconte une histoire évolutive ........................ 22. Construire un arbre avec Phylogène ................................................................................ 42.1 Cas des arbres construits à partir des comparaisons de molécules ........................... 42.2 Cas des arbres construits à partir d'une matrice taxons-caractères ........................... 43. Comparer les degrés de parenté ...................................................................................... 64. Placer l'apparition d'un caractère ..................................................................................... 75. Retrouver les caractères dérivés possédés par un taxon ................................................ 86. Placer un taxon dans un arbre phylogénétique ................................................................ 9 © Bill Watterson
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 2 1.L'arbrephylogénétique:undocumentquiraconteunehistoireévolutive • On repr ésente aujourd'hui les liens de parenté entre les espèces sous la f orme d'arbres phylogénétiques. Le mot phylo génétique vient de la phylogénie (du grec phullon : rameau, feuille) : l'histoire des différentes lignées d'êtres vivants actuels et fossiles. o L'arbre retrace une his toire évolutive. Le tronc rep résente la péri ode la plus ancienne. Les rameaux représentent des lignées dont les extrémités seulement sont les taxons actuels (le taxon est l'unité classée ; dans de nombreux arbres les taxons sont des espè ces, mais da ns certains a rbres on classe des group es d'espèces comme " lézards » " oiseaux » etc .). les taxons actuels son t donc placées sur une même ligne, à la cime de l'arbre (la ligne du présent). Les taxons fossiles sont tous en dessous de cette ligne. Plus le degré de parenté entre deux taxons est élevé, plus la séparation entre leurs lignées est tardive. Ils ont donc accumulé de nombreux caractères évolués (dérivés) communs et leur der nier ancêtre commun est récent. o L'arbre phylogénétique permet de reconstituer les ét apes qui conduisent à l'apparition des taxons actuels . Au fur et à mesure que l'on parc ourt le traj et évolutif qui conduit de la base du tronc jus qu'au t axon étudié, on franchi t différentes étapes de spéciation et on ac cumule des caractères dérivés (ou caractères évolués) successifs. o Chaque bifurcation de l'arbre correspond à un ancêtre, à partir duquel deux lignées se sont séparées. Les ancêtres sont toujours hypothétiques, c'est-à-dire qu'on ne peut identi fier avec certitude une espèce fossile comm e étant cet ancêtre, dont l'existence même n'est qu'une supposition raisonnée. Les lignées issues de cet ancêtre ont hérité de lui un ensemble de caractères dérivés que l'ancêtre est le premier à avoir possédé . Ainsi, cet ancêtre et tous ses descendants forment un clade, ou groupe monophylétique, qui est défini par cet ensemble de caractères dérivés, par exemple, le clade des mammifères regroupe
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 3 toutes les espèces actuelles ou fossiles ayant à la fois les caractères généraux des vertébrés avec en plus des poils et des mamelles. o Les clades d'un arbre sont emboîtés les uns dans les autres : ils sont hiérarchisés. Par exemple, le clade des vertébrés contient le clade des mammifères qui contient lui-même le clade des primates, c'est à dire des mammifères ayant une vision binoculaire, des ongles, et des pouces opposables aux autres doigts. Remarque : Les groupes de l'ancienne clas sif ication ne sont pas tous monophylétiques. Par exemple, le grou pe des Reptiles est dé fini à partir d 'états dérivés de caractères (hypapophyse : quille ventrale sous les vertèbres cervicales ; iris compre nant des mucles striés ; pr oduction d'acide ornithurique). Or ces caractères sont aussi présents chez les Oiseaux, alors que ceux-ci sont exclus des reptiles. Un tel groupe est appelé paraphylétique. Il est peu utile, voir e trompeur lorsqu'il s'agit de retracer les liens de parenté entre les taxons actuels.
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 4 2.Construireunarbreavecphylogène • Pour construi re un arbre phylogénétique à par tir d'un ensemble de taxons (ou collection), on commence par construire une matrice taxons-caractères qui permet de comparer ces taxons. Les caract ères utilisés comme critères de parenté doivent dépendre du programme génétique des êtres vivants. Pour cette raison, ce sont : o Les ressem blances morphologiques et anatomiques (établies sur de larges populations plutôt que sur des individus isolés qui peuvent être malformés ou atypiques). o Les ressemblances morphologiques et anatomiques embryonnaires. o Les ressemblances moléculaires portant sur la séquence des gènes ou protéines présents chez les espèces étudiées. 2.1Casdesarbresconstruitsàpartirdescomparaisonsdemolécules • Le degré de parenté entr e l es taxons corr espond à l'ensemble de leurs ressemblances, qu'elles portent sur des carac tères dérivés ou des caractère s ancestraux. Lorsque l'on compare la séquence d'un gène ou d'une protéine chez plusieurs taxons, plus le % de ressemblance entre les séquences étudiées est élevé, plus le degré de par enté est él evé. Cett e m éthode quantitative est appelée phénétique. Les conclusions qui se fondent sur cette méthode dépendent étroitement du choix plus ou moins judicieux des molécules choisies pour la comparaison et de l'ampleur de l'échantillon statistique utilisé.1 2.2Casdesarbresconstruitsàpartird'unematricetaxons-caractères • On comm ence par construire la matr ice taxons-caractères en cl iquant sur les taxons et les c aractères retenus, pui s on remplit la matrice à l'aide des donnée s sur chaque taxon fournies par le logiciel. • Le logiciel demande ensuite de polariser la matrice, c'est-à-dire de déterminer l 'état ancestral et l'état évolué (ou dérivé) de chaque caractère. Ce travail est facilité si l'on a inclu dans la matrice un extra-groupe (ici la lamproie), c' est-à-dire un taxon suppos é posséder l'ensemble des caractères étudiés à l'état primitif. 1 Remarque : dans certaines études moléculaires, il est possible de déterminer l'état ancestral de la séquence étudiée. Dans un tel cas, on peut utiliser les mutations ayant affecté la séquence originelle comme des caractères dérivés et classer les taxons selon la méthode cladistique exposée ici pour les caractères morpho-anatomiques.
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 5 • Une fois la matrice polarisée, le logiciel permet de construire l'arbre en créant les différents noeuds (ou bifurcations) de l'arbre. o Pour créer le s noeuds, il faut rapprocher d'abord les deux taxons qui partagent le plus grand nombre de caractères dérivés. o Puis chercher dans les t axons restants celui qui partag e le plus grand nombre d' états dérivés avec les taxons déjà rattachés et le rattacher à l' arbre en créant un second noeud plus ancien (placé plus bas dans l' arbre), et ainsi de suite. o Dans cette méthode, le degré de parenté entre deux espèces correspond à leur nombre de caractères dérivés communs; les caractères primitifs ne sont pas pris en compte, ni l'absence de tel ou tel caractère2. Cette méthode qui privilégie les états dérivés de caractères s'appelle la cladistique. • Lorsque plusieurs arbres sont possibles, on choisit celui qui implique le plus petit nombre de pas évolut ifs , c'est à dire le plus petit nombre de transfor mations, apparitions ou disparitions de caractères. C'est le principe de parcimonie. 2 Sauf dans le cas où cette absence est le résultat d'une perte d'un caractère ancestral
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 6 3.Comparerlesdegrésdeparenté• Pour comparer les liens de parenté entre les taxons, il faut prendre les taxons deux par deux et évaluer pour chaque paire de taxons le lien de parenté ; pour cela : • Identifier dans l'arbre le plus récent ancêtre commun aux deux taxons étudiés • Plus cet ancêtre est proche de la ligne du présent plus le lien de parenté entre les deux taxons est fort. • exemple: dans l'arbre proposé, le lien de parenté entre le Crapaud et le Chat ( 3 caractères dérivés communs, ancêtre 3) est plus fort que le lien de parenté entre le Crapaud et le Thon (2 caractères dérivés communs, ancêtre 2). L'ancêtre 3 est plus proche de la ligne du présent que l'ancêtre 2.
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 7 4.Placerl'apparitiond'uncaractère • Repérer l'état dérivé du caractère étudié dans la matrice polarisée : cet état dérivé résulte d'une innovation évolutive ou pas évolutif. • Repérer les taxons qui possèdent cet état dérivé et remonter dans l'arbre jusqu'au noeud qui correspond à leur plus récent ancêtre commun • L'apparition du caractère étudié doit êtr e placée sur le segm ent de tr onc qui se trouve immédiatement en-dessous de cet ancêtre • Exemple : l'Aigle et le Crocodile sont les seuls à posséder la fenêtre mandibulaire; elle est apparue dans la lignée de leur ancêtre commun (ancêtr e 6) qui leur a transmis ce caractère. Mai s elle n' est pas présente chez l'ancêt re 4, puisque plusieurs des descendants de cet ancêtre ne possèdent pas ce caractère. On place donc l'apparition de la fenêtre mandibulaire sur le segment de branche situé entre l'ancêtre 4 et l'ancêtre 6.
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 8 5.Retrouverlescaractèresdérivéspossédésparuntaxon • Retrouver les différents caractères dérivés possédés par un taxon revient à retrouver les différents pas évolutifs franchis par ce taxon au cours de son "chemin évolutif", c'est à dire au cours du chemin qui part de la base du tronc de l'arbre phylogénétique et qui conduit à ce taxon. Vous allez donc : • Placer sur l'arbre les apparitions de tous les caractères dérivés (voir section 4) • Partir du tronc de l'arbre et parcourir l'arbre de la base au sommet jusqu'au taxon étudié en notant tous les caractères apparus sur votre chemin. • Exemple le Croc odile est l'aboutissement d'u ne histoire évolutif marqu ée par l'acquisition successive des caractères suivant: vertèbres, mâchoires, doigts, amnios, fenêtre mandibulaire.
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 9 6.Placeruntaxondansunarbrephylogénétique • Repérer les caractères dérivés qui figurent dans l'arbre et sont présents dans ce taxon • Repérer parmi les taxons de l'arbre celui qui possède les mêmes caractères dérivés o Si l'un des taxons présente exactement les mêmes caractères dérivés, il suffit de créer une bifurcation dans la lignée propre à ce taxon (=sur la branche où il est tout seul) et de placer le nouveau taxon sur la nouvelle branche issue de la bifurcation. On veillera à aligner le taxon sur la ligne du présent s'il s'agit d'un espèce présente ou en-dessous s'il s'agit d'un espèce fossile. o Si aucu n taxon ne présen te exactement les mêmes ca ractères dérivés, placer d'abord toutes les apparitions de caractères sur l'arbre phylogénétique (voir section 4), puis parcourir l'arbre depuis le tronc jusqu'à l'ancêtre ayant acquis le dernier caractère dérivé possédé par le taxon à placer. Créer une bifurcation dans le segment de tronc qui se trouve immédiatement au-dessus de cet ancêtre et placer le nouveau taxon sur cette branche (sans oublier de vérifier s'il faut placer le nouveau taxo n sur la ligne du présent ou en-dessous). On peut aussi créer la nouvelle bifurcation directement à partir de l'ancêtre (depuis cet ancêtre partiront alors trois branches : les deux branches déjà présentes et celle que vous venez de créer) • Exemple : l'espèce fossile Purgatorius (ci-contre) possède des vertèbr es, une mâchoire, des doigts, un amn ios et un placenta mais ne possède pas d'ongles. L'ancêtre ayant acquis le der nier caractère dérivé possédé par Purgatorius (le placenta) est donc l'ancêt re 5. La bi furcation de purgatorius est donc placée sur le segment de l 'arbre situé immédiatement au-dessus entre l'ancêtre 5 et l'ancêtre 7. Purgatorius est une espèce fossile: il doit être placé en-dessous de la ligne du présent.
T. Leclerc - Comprendre et exploiter un arbre phylogénétique 10 FIN © Bill Watterson
quotesdbs_dbs17.pdfusesText_23[PDF] interprétation d'une médiane
[PDF] interprétation des coefficients de régression
[PDF] interprétation des essais cliniques pour la pratique médicale pdf
[PDF] interprétation des ratios d analyse financière
[PDF] interprétation des résultats bactériologiques
[PDF] interprétation du bfr en jours de ca
[PDF] interprétation du test de ljung box
[PDF] interpretation ebe
[PDF] interprétation économique du dual
[PDF] interprétation économique du multiplicateur de lagrange
[PDF] interprétation électrophorèse adn
[PDF] interprétation essai pressiométrique
[PDF] interprétation glm
[PDF] interprétation graphique second degré
