 Shared Protocol - Extracting DNA using Phenol-Chloroform
Shared Protocol - Extracting DNA using Phenol-Chloroform
Extracting DNA Using Phenol-Chloroform. Reagents Needed. 1. Phenol/chloroform/isoamyl alcohol (PCI) solution (25:24:1) DNase (RNase- and Protease-Free -.
 Optimisation dun protocole dextraction et de purification dADN de
Optimisation dun protocole dextraction et de purification dADN de
Standard methods of DNA purification with phenol and chloroform were tested. The optimal conditions of extraction and purification are proposed to obtain pure
 PROTOCOL OF DNA EXTRACTION 1- Phenol/chloroform extraction
PROTOCOL OF DNA EXTRACTION 1- Phenol/chloroform extraction
PROTOCOL OF DNA EXTRACTION. 1-. Phenol/chloroform extraction and ethanol precipitation. (INPBARCELONA). 1.1 Phenol extraction of Genomic DNA from frozen
 Phenol/Chloroform Extraction and Ethanol Precipitation
Phenol/Chloroform Extraction and Ethanol Precipitation
Dissolve in Water nuclease-free or TE buffer. Note. Use Glycogen to maximize the yield of DNA during precipitation. This protocol is for the Phenol/Chloroform
 Dr. ZIADA-BOUCHAAR H. MI Génétique moléculaire Université
Dr. ZIADA-BOUCHAAR H. MI Génétique moléculaire Université
Différents protocoles pour extraire l'ADN avec même schéma de principe : Les 1- Méthode au phénol-chloroforme : le principe consiste à traiter le lysat ...
 Optimalisation de lextraction dADN génomique de la morelle jaune
Optimalisation de lextraction dADN génomique de la morelle jaune
Paramètres testés pour les étapes importantes de l'extraction de l'ADN en référence au protocole de Phénol/chloroforme/alcool isoamylique (25:24:1) &.
 Technical note: Preparing DNA for PacBio HiFi sequencing
Technical note: Preparing DNA for PacBio HiFi sequencing
See www.extractdnaforpacbio.com as a resource database of customer protocols for DNA extraction phenol/chloroform. These chemicals are strong oxidizers and ...
 08.02.005 f2.0 Extraction de lADN à partir du sang Procédure
08.02.005 f2.0 Extraction de lADN à partir du sang Procédure
26 juin 2012 7.1.1 Traiter le sang total comme potentiellement infectieux. 7.1.2 Le phénol et le chloroforme sont des solvants organiques et doivent être ...
 Evaluation of different methods for DNA extraction from milk
Evaluation of different methods for DNA extraction from milk
28 févr. 2014 The protocol for classical phenol-chloroform extraction was based on that described by DE et al. [5]. Briefly 1 ml milk sample was centrifuged ...
 Shared Protocol - Extracting DNA using Phenol-Chloroform
Shared Protocol - Extracting DNA using Phenol-Chloroform
Extracting DNA Using Phenol-Chloroform. Reagents Needed. 1. Phenol/chloroform/isoamyl alcohol (PCI) solution (25:24:1) DNase (RNase- and Protease-Free -.
 Optimisation dun protocole dextraction et de purification dADN de
Optimisation dun protocole dextraction et de purification dADN de
Standard methods of DNA purification with phenol and chloroform were tested. The optimal conditions of extraction and purification are proposed to obtain pure
 Protocole dextraction
Protocole dextraction
EXTRACTION DE L'ADN A PARTIR DE LEUCOCYTES ISOLES (*Thermus aquaticus) par la méthode conventionnelle au Phénol-Chloroforme. Protocole d'extraction.
 Protocole dextraction dADN de Pseudomonas aeruginosa La
Protocole dextraction dADN de Pseudomonas aeruginosa La
Dans certains cas il peut être utilisé pour une PCR après dilution au 1/50 dans de l'eau. Extraction au phénol/chloroforme. ?Traitement au CTAB. Pour des
 Optimalisation de lextraction dADN génomique de la morelle jaune
Optimalisation de lextraction dADN génomique de la morelle jaune
phénol/chloroforme/alcool isoamylique 25:24:1 sont ajoutés au surnageant l'ensemble est agité
 Compte-rendu de stage méthodologique
Compte-rendu de stage méthodologique
et la qualité de l'ADN extrait ainsi que la diversité obtenue. I. Le choix des protocoles d'extraction. Extraction au phénol-chloroforme :.
 VETAGRO SUP
VETAGRO SUP
L'extraction au phénol-chloroforme la technique de référence . Protocole EPICENTRE
 Protocol for Phage DNA Extraction with Phenol:Chloroform
Protocol for Phage DNA Extraction with Phenol:Chloroform
21 sept. 2018 Protocol for Phage DNA Extraction with Phenol:Chloroform ... In this protocol you extract the genomic DNA from the phages in a lysate.
 08.02.005 f2.0 Extraction de lADN à partir du sang Procédure
08.02.005 f2.0 Extraction de lADN à partir du sang Procédure
1 juin 2012 des acides nucléiques (ADN et ARN) dérivés de ces échantillons. ... méthode au phénol/chloroforme (solvant organique). 2.0 PORTÉE.
 Ministère de lEnseignement Supérieur de la Recherche
Ministère de lEnseignement Supérieur de la Recherche
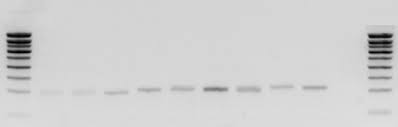
Figure 10: Gel de qualité de F ADN génomique de Liposcelis tricolor (5 individus) avec le protocole d'extraction sans phénol chloroforme.
 [PDF] Shared Protocol - Extracting DNA using Phenol-Chloroform - PacBio
[PDF] Shared Protocol - Extracting DNA using Phenol-Chloroform - PacBio
Extracting DNA Using Phenol-Chloroform Reagents Needed 1 Phenol/chloroform/isoamyl alcohol (PCI) solution (25:24:1) DNase (RNase- and Protease-Free -
 [PDF] Extraction de lADN - Bibliomer
[PDF] Extraction de lADN - Bibliomer
L'extraction de l'ADN (acide désoxyribonucléique) est une technique qui isole de l'ADN à partir d'une cellule en Du phénol et du chloroforme sont par
 [PDF] Optimisation dun protocole dextraction et de purification dADN de
[PDF] Optimisation dun protocole dextraction et de purification dADN de
Standard methods of DNA purification with phenol and chloroform were tested The optimal conditions of extraction and purification are proposed to obtain pure
 [PDF] Méthodes dextraction et de purification du matériel génétiques
[PDF] Méthodes dextraction et de purification du matériel génétiques
Différents protocoles pour extraire l'ADN avec même schéma de principe : Les principaux procédés d'extraction Le traitement au phénol chloroforme (CHCL
 [PDF] Phenol/chloroform extraction and ethanol precipitation
[PDF] Phenol/chloroform extraction and ethanol precipitation
PROTOCOL OF DNA EXTRACTION 1- Phenol/chloroform extraction and ethanol precipitation (INPBARCELONA) 1 1 Phenol extraction of Genomic DNA from frozen
 [PDF] 0802005 f20 Extraction de lADN à partir du sang - CTRNet
[PDF] 0802005 f20 Extraction de lADN à partir du sang - CTRNet
26 jui 2012 · Le but de ce document est de définir les procédures normalisées à suivre pour les banques du RCBT lors de l'extraction de l'ADN des cellules
 [PDF] Phenol-chloroform extraction - ResearchGate
[PDF] Phenol-chloroform extraction - ResearchGate
A phenol-chloroform extraction is a liquid-liquid extraction Protocol for Isolation of RNA from Animal Cells or Tissues Materials Reagents
 [PDF] Extraction dADN génomique bactérien
[PDF] Extraction dADN génomique bactérien
L'objectif de la séance est de purifier de l'ADN génomique d'E coli Procéder à une nouvelle extraction par le mélange phénol/chloroforme :
 [PDF] 41072949pdf - International Nuclear Information System (INIS)
[PDF] 41072949pdf - International Nuclear Information System (INIS)
Deux différents protocoles d'extraction de l'ADN génomique sont employés l'extraction i phénol chloroforme TBaruffi et al 1995) et sans le phénol chloroforme
 [PDF] Protocol for Phage DNA Extraction with Phenol:Chloroform
[PDF] Protocol for Phage DNA Extraction with Phenol:Chloroform
21 sept 2018 · Protocol for Phage DNA Extraction with Phenol:Chloroform In this protocol you extract the genomic DNA from the phages in a lysate
Pourquoi nous avons choisi la méthode d'extraction au chloroforme ?
L'avantage de la méthode au phénol-chloroforme est la simplicité du protocole, la dénaturation rapide des nucléases et la stabilisation des acides nucléiques.Quelles sont les étapes de l'extraction de l'ADN ?
Les méthodes d'extraction des acides nucléiques peuvent être classées en deux types différents :
Les méthodes en solution (comme la méthode au phénol-chloroforme)Les méthodes basées sur la phase solide (comme les billes magnétiques ou les colonnes de centrifugation).Quel est le rôle du phénol-chloroforme ?
Le phénol est un excellent agent dénaturant des protéines et il permet de séparer efficacement les protéines et les acides nucléiques. Il est ensuite éliminé par l'extraction avec du chloroforme (non miscible avec l'eau). La séparation des phases aqueuse et organique peut se faire par centrifugation.- L'extraction au phénol est une méthode couramment utilisée pour éliminer les protéines d'un échantillon d'ADN, par ex. éliminer les protéines du lysat cellulaire lors de la préparation de l'ADN génomique.
Center for Phage Technology Protocol: Phage DNA extraction - traditional
Texas A&M University, College Station, TX 77843 Rev. 9/21/20181 of 2
1 of 2
In this protocol you extract the genomic DNA from the phages in a lysate. The lysates are "dirty" in that they contain spent media components, cell wall debris, flagella, nucleic acids, bacterial proteins and unassembled phage proteins in addition to the phage themselves. There are many methods for purifying phage DNA from the rest of the lysate, This protocol is recommended specifically for phages that have not resulting in high-yield or high-quality DNA from a kit (like the Promega Wizard DNA Clean-up Kit used in the DNA Extraction Protocol). For best results, adhere to the recommendations for incubation times listed in each step.Day 1: PEG Precipitation
1. Take 1 volume of phage high titer lysate (see below) and add PEG (10% PEG-
8000, 1 M NaCl final concentration) at a ratio of 1:2 precipitant:lysate. Mix gently
by inversion. a. As a rule of thumb, 1 ml of phage lysate with a titer of 1 x 1010 contains about 0.5 μg of phage DNA, assuming the phage has a 50 kb genome. Typically 10 ml of a high-titer phage lysate is used for DNA extraction. More or less lysate (up to 20 ml) may be used depending on the phage stock titer and the expected genome size of the phage. For low-titer phage stocks, up to 20 ml of lysate may be used. With high titer stocks of large- genome phages, use 10 ml of lysate or less.2. Incubate at 4°C overnight (this is ideal, can also do 60 minutes on ice if you need
to work faster). Most phages are stable like this for several days.Day 2: DNA Extraction
1. Centrifuge the PEG-precipitated sample at 10,000 xg for 30 minutes.
2. Use 5mM MgSO
4 to re-suspend the pellet, pipetting gently up and down. AVOID introducing BUBBLES during the resuspension. Be sure to rinse down the sides of the tube to obtain all of the pellet. Transfer to a labeled 2 mL epi tube.3. Using 500 µl of the concentrated sample: add 1.25 ul of DNaseI and RNase (20
mg/ml) and incubate at 37 oC for 1hour.
a. (You can scale all volumes up if more than 500 uL are needed for resuspension).4. Add 1.25 µl of Proteinase K 20 mg/ml stock (20µg total) and 25 µl 10% SDS
stock (0.5% final concentration) and 20 ul of 0.5 M EDTA pH 8.0 (20 mM final).Mix and incubate 1 hour at 60°C.
5. Allow to cool to room temperature.
6. Add an equal volume of phenol:chloroform (1:1) and invert several times.
7. Spin 3000 xg (6000 rpm on microfuge), 5 minutes room temperature.
8. Carefully transfer the supernatant with a wide-bore pipette tip to a fresh, labeled
2 mL epi tube.
9. Add an equal volume of phenol:choroform (1:1), invert.
Protocol for Phage DNA Extraction with Phenol:ChloroformCenter for Phage Technology Protocol: Phage DNA extraction - traditional
Texas A&M University, College Station, TX 77843 Rev. 9/21/20182 of 2
2 of 2
10. Centrifuge as above and transfer the supernatant again.
11. Add an equal volume of chloroform, invert.
12. Centrifuge as above and transfer the supernatant to a fresh, labeled 2mL epi
tube. a. Depending on volume here you may need to use a 15mL falcon tube13. Add 1/10 volume of 3 M NaOAc (pH 7.5), and 2.5 volumes of ice cold ethanol
(100%). Mix well and incubate at -20°C overnight. a. A fast method allows incubation on ice for 15-30 minutes.Day: 3 DNA Precipitation
1. Centrifuge in a benchtop microfuge at max speed 20 minutes (10000 rpm for
15ml tubes as tube rating allows, 15000 rpm on the microfuge).
2. Carefully remove supernatant and fill tube halfway with 70% ethanol (made from
100% with purified nuclease free water), spin at max speed for 2 minutes.
3. Repeat the above step one time (2nd 70% wash).
4. Remove as much ethanol as possible without disturbing the pellet - good idea to
hold onto the supernatant until DNA recovery has been confirmed.5. Leave tube open on bench ~ 15-30 minutes to let ethanol disperse.
6. Dissolve in TE buffer (~ pH 7.6).
quotesdbs_dbs7.pdfusesText_13[PDF] force d'inertie d'entrainement
[PDF] sailing terms english french
[PDF] les fleurs du mal figure de style titre
[PDF] que symbolise l'albatros
[PDF] procédé stylistique le pin des landes
[PDF] l'albatros analyse figure de style
[PDF] exercice corrigé de calcul de moment d inertie
[PDF] fonction du poète baudelaire l albatros
[PDF] la phrase traduction en espagnol
[PDF] prince des nuées
[PDF] exercices corrigés physique seconde forces et principe d inertie
[PDF] eduscol tpe 2018
[PDF] spleen lecture analytique
[PDF] lecture analytique le crapaud
