 Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Microbiologie Moléculaire des Actinobactéries. ICI. Microbiologie ; Métabolisme ; Biologie de synthèse ;. Antibiotiques ; Génomique ; Dynamique de la
 METABOLISME ET NUTRITION BACTERIENS.pdf
METABOLISME ET NUTRITION BACTERIENS.pdf
métabolisme respiratoire soit le NAD+ l'oxydation complète du glucose par la voie aérobie du cycle tricarboxylique correspond à la réaction globale
 Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Microbiologie ; Fonctions des enzymes ; Réseaux métaboliques. Bioinformatics Microbiologie ; Métabolisme ; Biologie de synthèse ;. Antibiotiques ...
 Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Microbiologie Moléculaire des Actinobactéries. ICI. Microbiologie ; Métabolisme ; Biologie de synthèse ;. Antibiotiques ; Génomique ; Dynamique de la
 Présentation PowerPoint
Présentation PowerPoint
17 janv. 2022 Métabolisme (INRAe). • RPE. • RMN (500 400
 Corrigé TD Biologie appliquée Microbiologie Nutrition Alimentation
Corrigé TD Biologie appliquée Microbiologie Nutrition Alimentation
PARTIE 2 : MICROBIOLOGIE En effet si les dépenses énergétiques ne sont pas augmentées
 Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Microbiologie Moléculaire des Actinobactéries. ICI. Microbiologie ; Métabolisme ; Biologie de synthèse ;. Antibiotiques ; Génomique ; Dynamique de la
 Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Microbiologie Moléculaire des Actinobactéries. ICI. Microbiologie ; Métabolisme ; Biologie de synthèse ;. Antibiotiques ; Génomique ; Dynamique de la
 Untitled
Untitled
2.1 - Spécificité du monde vivant : Organisme; espèce; microbiologie; métabolisme; reproduction et hérédité; macromolécules. 2.2 - Santé publique
 Master Mention Sciences du vivant Spécialité Biologie des micro
Master Mention Sciences du vivant Spécialité Biologie des micro
microbiologie. Cette formation permet aux étudiants d'élaborer des projets métabolisme inorganique. Page 6. Objectifs en termes de compétences.
 Cours de Biochimie Microbienne L3
Cours de Biochimie Microbienne L3
Département de Microbiologie. Cours de. Biochimie Microbienne L3 Réactions permettant la fourniture de l'énergie nécessaire : métabolisme énergétique.
 METABOLISME ET NUTRITION BACTERIENS.pdf
METABOLISME ET NUTRITION BACTERIENS.pdf
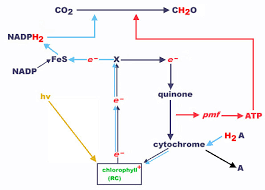
Le métabolisme est l'ensemble des réactions biochimiques mises en jeu par un organisme pour permettre sa croissance (figure 1). Les réactions métaboliques
 Travaux pratiques de microbiologie générale
Travaux pratiques de microbiologie générale
Laboratoire de Microbiologie Appliquée L'eau est nécessaire au métabolisme bactérien les milieux de culture doivent en contenir.
 Master Microbiologie et contrôle de qualité
Master Microbiologie et contrôle de qualité
axes multiples tels que : Métabolisme des procaryotes Microbiologie des aliments : aspect technologique
 Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Laboratoire Nom de léquipe Site internet de léquipe Mots-clés
Microbiologie ; Fonctions des enzymes ; Réseaux métaboliques. Bioinformatics; Comparative genomics; Microbiologie ; Métabolisme ; Biologie de synthèse ;.
 Chapitre 3: Nutrition bactérienne et Types trophiques
Chapitre 3: Nutrition bactérienne et Types trophiques
Métabolisme bactérien. Mme : BELKHIRI Farida (MCB). Microbiologie en 2019/2020 (i) l'état végétatif avec une grande activité métabolique.
 Etude de la diversité métabolique des micro-organismes des
Etude de la diversité métabolique des micro-organismes des
Je voudrais exprimer mes plus vifs remerciements à Françoise Lesongeur qui m'a fait partager ses connaissances en microbiologie et biologie moléculaire. Merci
 Chercheur(e) spécialiste du microbiote et du métabolisme procaryote
Chercheur(e) spécialiste du microbiote et du métabolisme procaryote
Le candidat sélectionné sera affilié au Département de microbiologie infectiologie et immunologie de la. Faculté de médecine. Nous recherchons un(e)
 BIOCHIMIE MICROBIENNE
BIOCHIMIE MICROBIENNE
Département de Microbiologie Métabolisme des produits minéraux. ... biochimique le terme respiration
 Master Mention Sciences du vivant Spécialité Biologie des micro
Master Mention Sciences du vivant Spécialité Biologie des micro
master BMO parcours microbiologie
 [PDF] METABOLISME ET NUTRITION BACTERIENS
[PDF] METABOLISME ET NUTRITION BACTERIENS
Le métabolisme est l'ensemble des réactions biochimiques mises en jeu par un organisme pour permettre sa croissance (figure 1) Les réactions métaboliques
 [PDF] Métabolisme Biochimique
[PDF] Métabolisme Biochimique
Métabolisme Biochimique Données Thermodynamiques 1 Les deux principes de thermodynamique Thermodynamique: analyse énergétique au sein d'un système;
 [PDF] Ecologie microbienne et métabolisme associé : étude de leau
[PDF] Ecologie microbienne et métabolisme associé : étude de leau
Ce présent travail porte sur l'étude de la microbiologie d'un environnement profond constitué par une couche argileuse datée d'environ 160 millions d'années et
 [PDF] Cours de Microbiologie Générale
[PDF] Cours de Microbiologie Générale
%2520Nutrition%2520bact%25C3%25A9rienne.pdf
 Bacterio19-Metabolisme Biochimique Bacterien PDF - Scribd
Bacterio19-Metabolisme Biochimique Bacterien PDF - Scribd
Le métabolisme énergétique d'une bactérie chimioorganotrophe Cours Microbiologie Alimentaire BOUBENDIR Abdelhafid CUM 2014 PDF (1)
 (PDF) Le Métabolisme Bactérien Djazou Microbio - Academiaedu
(PDF) Le Métabolisme Bactérien Djazou Microbio - Academiaedu
Le Métabolisme Bactérien Download Free PDF Pour une compréhension claire des principes de biochimie et de biologie moléculaire en relation avec la
 [PDF] COURS DE MICROBIOLOGIE GENERALE
[PDF] COURS DE MICROBIOLOGIE GENERALE
Le métabolisme est l'ensemble des réactions biochimiques mises en jeu par un organisme pour permettre sa croissance (figure 1) Les réactions métaboliques
 Physiologie bactérienne : Métabolisme et Applications
Physiologie bactérienne : Métabolisme et Applications
Physiologie bactérienne: Nutrition et Croissance :métabolisme bactérienmétabolisme de glucide métabolisme énergétique cycle de krebsBIOCHIMIQUE
 [PDF] Physiologie des microorganismes - MICROBIOLOGIE -
[PDF] Physiologie des microorganismes - MICROBIOLOGIE -
Certains micro-organismes sont capables d'assimiler de nombreuses substances organiques différentes tandis que d'autres ont des capacités métaboliques
 [PDF] Catabolisme des glucides - Faculté
[PDF] Catabolisme des glucides - Faculté
Licence Microbiologie Appliquée Mme Djouahra-Fahem Chapitre 2 : Catabolisme des Glucides Destinée du pyruvate 1- Métabolisme aérobie du pyruvate
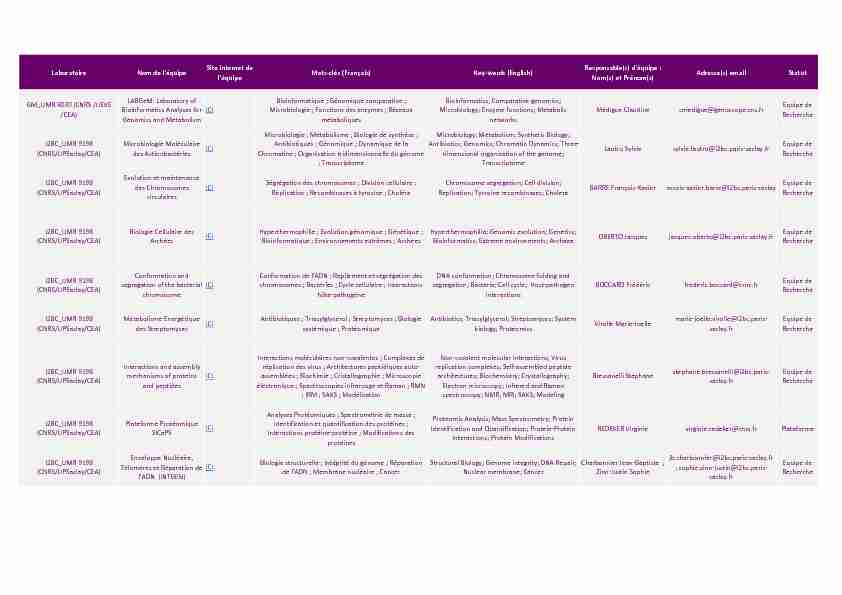
LaboratoireNom de l'équipeSite internet de
l'équipeMots-clés (Français)Key-words (English)Responsable(s) d'équipe :Nom(s) et Prénom(s)Adresse(s) emailStatut
GM_UMR 8030 (CNRS /UEVE
/CEA)LABGeM: Laboratory of
Bioinformatics Analyses for
Genomics and Metabolism
ICIBioinformatique ; Génomique comparative ;
Microbiologie ; Fonctions des enzymes ; Réseaux métaboliquesBioinformatics; Comparative genomics;
Microbiology; Enzyme functions; Metabolic
networks Médigue Claudine cmedigue@genoscope.cns.frEquipe deRecherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Microbiologie Moléculaire
des ActinobactériesICI Microbiologie ; Métabolisme ; Biologie de synthèse ;Antibiotiques ; Génomique ; Dynamique de la
Chromatine ; Organisation tridimensionnelle du génome ; TranscriptomeMicrobiology; Metabolism; Synthetic Biology;
Antibiotics; Genomics; Chromatin Dynamics; Three-
dimensional organization of the genome;Transcriptome
Lautru Sylvie sylvie.lautru@i2bc.paris-saclay.frEquipe deRecherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Evolution et maintenance
des Chromosomes circulaires ICISégrégation des chromosomes ; Division cellulaire ; Réplication ; Recombinases à tyrosine ; CholéraChromosome segregation; Cell division;
Replication; Tyrosine recombinases; CholeraBARRE François-Xavierfrancois-xavier.barre@i2bc.paris-saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Biologie Cellulaire des
ArchéesICIHyperthermophilie ; Evolution génomique ; Génétique ; Bioinformatique ; Environnements extrêmes ; ArchéesHyperthermophilia; Genomic evolution; Genetics;
Bioinformatics; Extreme environments; ArchaeaOBERTO Jacques jacques.oberto@i2bc.paris-saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Conformation and
segragation of the bacterial chromosome ICI Conformation de l'ADN ; Repliement et ségrégation des chromosomes ; Bactéries ; Cycle cellulaire ; Interactions hôte-pathogèneDNA conformation; Chromosome folding and
segregation; Bacteria; Cell cycle; Host-pathogen interactions BOCCARD Frédéric frederic.boccard@cnrs.frEquipe deRecherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Métabolisme Energétique
des StreptomycesICIAntibiotiques ; Triacylglycérol ; Streptomyces ; Biologie systémique ; Protéomique Antibiotics; Triacylglycerol; Streptomyces; System biology; ProteomicsVirolle Marie-Joelle marie-joelle.virolle@i2bc.paris- saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Interactions and assembly
mechanisms of proteins and peptides ICI Interactions moléculaires non-covalentes ; Complexes de réplication des virus ; Architectures peptidiques auto- assemblées ; Biochimie ; Cristallographie ; Microscopie électronique ; Spectroscopies infrarouge et Raman ; RMN ; IRM ; SAXS ; ModélisationNon-covalent molecular interactions; Virus
replication complexes; Self-assembled peptide architectures; Biochemistry; Crystallography;Electron microscopy; Infrared and Raman
spectroscopy; NMR; MRI; SAXS; Modeling Bressanelli Stéphane stephane.bressanelli@i2bc.paris- saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Plateforme Protéomique
SICaPSICI
Analyses Protéomiques ; Spectrométrie de masse ; Identification et quantification des protéines ; Interactions protéine-protéine ; Modifications des protéinesProteomic Analysis; Mass Spectrometry; Protein
Identification and Quantification; Protein-ProteinInteractions; Protein Modifications
REDEKER Virginie virginie.redeker@cnrs.frPlateformeI2BC_UMR 9198
(CNRS/UPSaclay/CEA)Enveloppe Nucléaire,
Télomères et Réparation de
ICIBiologie structurelle ; Intégrité du génome ; Réparation de l'ADN ; Membrane nucléaire ; CancerStructural Biology; Genome integrity; DNA Repair;
Nuclear membrane; Cancer
Charbonnier Jean-Baptiste ;
Zinn-Justin Sophie
jb.charbonnier@i2bc.paris-saclay.fr ; sophie.zinn-justin@i2bc.paris- saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Stress Oxydant et
DétoxicationICI
Enzymologie ; Résonance Paramagnétique Electronique ; Raman ; Modélisation moléculaire ; Hémoprotéines ;Cytochrome P450 (CYP) ; NO Synthase (NOS)
Oxidative Stress; Reactive Nitrogen and Oxygen
Species (RNOS); Structure-Function Relationship;
Enzymology; Electronic Paramagnetic Resonance;
Raman; Molecular Modeling; Hemoproteins;
Cytochrome P450 (CYP); NO Synthase (NOS)
Santolini Jérôme jerome.santolini@cea.frEquipe deRecherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Laboratory of Membrane
Proteins and Membrane
Systems
ICIProtéines membranaires ; Membranes ; Mécanismes moléculaires ; Maladies ; Homéostasie lipidiqueMembrane proteins; Membranes; Molecular
mechanisms; Diseases; Lipid homeostasisLenoir Guillaume ; Vázquez-
Ibar José Luis
guillaume.lenoir@i2bc.paris- saclay.fr ; jose-luis.vazquez- ibar@i2bc.paris-saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Assemblage moléculaire et
intégrité du génome (AMIG) ICIBiologie structurale ; Cancer ; Bioinformatique ; Design ; "Docking" Structural biology; Cancer; Bioinformatics; Design;Docking
Guerois Raphael ;
Ochsenbein Françoise
guerois@cea.fr ; francoise.ochsenbein@cea.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Plateforme RMNICI Structure tridimensionnelle ; Interaction ; Protéines intrinsèquement dépliées ; Modification post- traductionnelles ; DynamiqueThree-dimensional structure; Interaction;
Inherently unfolded proteins; Post-translational
modification; DynamicsZINN-JUSTIN Sophiesophie.zinn@cea.fr ;
Francoise.OCHSENBEIN@cea.frPlateforme
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Structural Biochemistry of
Microtubules, Kinesins and
their Cargos ICIBiologie structurale ; Moteurs moléculaires ; Trafic intracellulaire ; Cycle cellulaire ; Cytosquelette Structural biology; Molecular motors; Intracellular trafficking; Cell cycle; CytoskeletonMénétrey Julie ; Gigant
Benoît
julie.menetrey@i2bc.paris-saclay.fr ; benoit.gigant@i2bc.paris-saclay.frEquipe de
Recherche
I2BC_UMR 9198
(CNRS/UPSaclay/CEA)Microbiologie et
enzymologie structurale (MESB3S) ICICristallographie des protéines; Interactions protéine- ligand ; Mesures d'affinité ; Modélisation ; Structure 3DProtein crystallography; Protein-ligand
interactions; Affinity measurements; Modeling; 3D structure MORERA Solange solange.morera@i2bc.paris-saclay.frEquipe deRecherche
ICSN -UPR2301
(CNRS/INSERM/INRAE/Pasteur)Spectrométrie de MasseICIChimie structurale ; Chimie analytique ; Métabolomique ;
Lipidomique
Structural chemistry; Analytical chemistry;
Metabolomics; LipidomicsTouboul David david.touboul@cnrs.frEquipe deRecherche
LBPA_UMR 8113 (ENS Paris-
Saclay/CNRS)
Structures et Interactions
des Acides NucléiquesICIARN rétroviraux ; Hélicases ; Protéine de nucléocapsides rétrovirales ; G-quadruplexes ; Biologie structurale Retroviral RNA; Helicases; Retroviral NucleocapsidProtein; G-quadruplexes; Structural biologyMAUFFRET Olivier olivier.mauffret@ens-paris-saclay.frEquipe de
Recherche
MICALIS (INRAE/AgroParisTech)
Plateforme d'Analyse
Protéomique de Paris Sud-
Ouest (PAPPSO)
ICIProtéomique ; Spectrométrie de masse ; Peptide; Quantification ; Modifications post-traductionnellesProteomics; Mass spectrometry; Peptide;
Quantification; Post-translational modificationsMonnet Véronique veronique.monnet@inrae.frPlateforme
SABNP_UMR-S 1204
(UPSaclay/INSERM)Laboratoire Structure et
Activité des Biomolécules
Normales et Pathologiques
(SABNP) ICI Dynamique et la structure de complexes ARN:protéine ; Contrôle de la traduction ; Régulation de la dynamique des microtubulesDynamics and structure of RNA:protein
complexes; Translation control; Regulation of microtubule dynamics Pastré David david.pastre@univ-evry.frPlateforme2I_UMR 1173 (INSERM/UVSQ)Inflammation chronique et
réponse immunitaire (IRIS)ICIImmunologie ; Génétique ; Génomique ; Epigénétique;Arthrite
Immunology; Genetics; Genomics; Epigenetics;
ArthritisBREBAN Maximemaxime.breban@aphp.frPlateformeBIOGER_UMR 1290
(INRAE/AgroParisTech)Plateau BioinformatiqueICIBioinformatique; Génomique; Transcriptomics; Gestion
de données; Base de données management;Databases; Lapalu Nicolasnicolas.lapalu@inrae.frPlateforme /
Plateau
techniqueEGCE_UMR 9191
(CNRS/UPSaclay)Pôle GénomeICI Génomique évolutive ; Conflits génétiques ; Biologie computationnelle ; Transfert horizontal ; Génétique des populationsEvolutionary genomics; Genetic conflict;
Computational biology; Horizontal transfer;
Population genetics
POLLET Nicolas Pollet ; HUA-
VAN Aurélie
Nicolas.Pollet@egce.cnrs-gif.fr ;
Aurelie.Hua-Van@egce.cnrs-gif.fr
Equipe de
Recherche
ESE_UMR 8079
(UPSaclay/CNRS/AgroParisTech)Diversité, Ecologie et
Evolution Microbiennes
(DEEM) ICIDiversité microbienne ; Evolution microbienne; Ecologie microbienne ; Phylogénomique ; Métagénomique Microbial diversity; Microbial evolution; Microbial ecology; Phylogenomics; MetagenomicsLopez-Garcia Purificacion puri.lopez@universite-paris-saclay.frEquipe de
Recherche &
Plateforme
FLUO (dont IMNC_UMR8165
CNRS/UPSaclay/Université Paris-
Diderot)
Modélisation des systèmes
BiologiquesICIModélisation mathématique ; Physique statistique ;Simulations numériques ; Cancer ; Gliomes
Mathematical modelling; Statistical physics;
Numerical simulations; Cancer; GliomasBadoual Mathilde badoual@imnc.in2p3.frEquipe deRecherche
GABI_UMR 1313
(INRAE/AgroParisTech)Biologie Intégrative et
Génétique Équine (BIGE)ICICheval ; Génomique; Myopathie ; Mélanome ;Microbiote
Horse; Genomics; Myoapthia; Melanoma;
MicrobiotaBarrey Eric eric.barrey@inrae.frEquipe deRecherche
GABI_UMR 1313
(INRAE/AgroParisTech)Génomique Biodiversité
Bioinformatique Statistique
(GiBBS) ICIDiversité génétique ; Conservation ; Volaille ; Statistique ;Bioinformatique
Genetic diversity; Conservation; Poultry; Statistics; BioinformaticsLaloë Denisdenis laloe@inrae.frEquipe deRecherche
GABI_UMR 1313
(INRAE/AgroParisTech)Génétique et AquacultureICIGénétique ; Poisson ; Sélection ; Physiologie ;
AquacultureGenetics; Fish; Breeding; Physiology; AquacultureDupont-Nivet Mathilde mathilde.dupont-nivet@inrae.frEquipe de
Recherche
GABI_UMR 1313
(INRAE/AgroParisTech)Génétique et Génomique
Bovine (G2B)ICIGénétique bovine ; Génomique ; BioinformatiqueBovine genetics; Genomics; BioinformaticsBoichard Didier didier.boichard@inrae.frEquipe de
Recherche
GenHotel_EA 3886 (UEVE)
Laboratoire Européen de
Recherche pour la
Polyarthrite Rhumatoïde
ICIGénétique humaine ; Génomique ; Statistique ; Biologie computationelle des systèmesHuman genetics; Genomics; Statistics;
Computational systems biologyPetit-Teixeira Elisabeth elisabeth.teixeira@univ-evry.frEquipe deRecherche
GM_UMR 8030 (CNRS /UEVE
/CEA)Analyses génomiques des
Eucaryotes (LAGE)ICI
Génomique des écosystèmes ; Métagenomique ; Génomique microbienne ; Génomique comparative ;Métatranscriptomique
Ecosystem genomics; Metagenomics; Microbial
genomics; Comparative genomics;Metatranscriptomics
Wincker Patrick pwincker@genoscope.cns.frEquipe deRecherche
GM_UMR 8030 (CNRS /UEVE
/CEA)Équipe R&D bio-
informatique et sequençage (LBGB) ICIBio-informatique ; Génomique ; Assemblage de novo ;Annotation de génomes ; Séquençage
Bioinformatics; Genomics; De novo assembly;
Genome annotation; SequencingAURY Jean-Marc jmaury@genoscope.cns.frEquipe deRecherche
GQE_UMR 0320, UMR 8120
(INRAE, UPSaclay, CNRS,AgroParisTech)
Structure et Evolution des
Chromosomes Fongiques
(SECF) ICIGénomique comparée ; Génomique fonctionnelle ; Levures pathogènes ; Levures ; Reproduction sexuéeComparative genomics; Functional genomics;
Pathogenic yeasts; Yeasts; Sexual reproductionFairhead Cécile cecile.fairhead@universite-paris- saclay.frEquipe de
Recherche
GQE_UMR 0320, UMR 8120
(INRAE, UPSaclay, CNRS,AgroParisTech)
Diversité, Evolution et
Adaptation des Populations
(DEAP) ICI Biodiversité des cultures ; Sélection participative ; Interactions plantes-plantes ; Génétique quantitative ;Modélisation
Crop biodiversity; Participatory breeding; Plant-
plant interactions; Quantitative genetics;Modelling
Enjalbert Jérôme jerome.enjalbert@inrae.frEquipe deRecherche
GQE_UMR 0320, UMR 8120
(INRAE, UPSaclay, CNRS,quotesdbs_dbs29.pdfusesText_35[PDF] métabolisme glucidique bactérien
[PDF] phosphorylation oxydative ppt
[PDF] ascorbate chaine respiratoire
[PDF] phosphorylation oxydative animation
[PDF] phosphorylation oxydative schéma
[PDF] phosphorylation oxydative cours
[PDF] phosphorylation oxydative pdf
[PDF] boucle microbienne milieu aquatique
[PDF] boucle microbienne définition
[PDF] examen chaine de markov corrigé
[PDF] processus de markov pour les nuls
[PDF] temperature pdf
[PDF] la chambre des officiers résumé film
[PDF] la chambre des officiers questionnaire reponse
