 Guide de validation des méthodes danalyses
Guide de validation des méthodes danalyses
28 oct. 2015 quantitatives). 7.1.1.2 Tableau de contingence. Le calcul de certaines caractéristiques de performance des méthodes qualitatives fait appel ...
 La Quantification Relative
La Quantification Relative
quantitative PCR: a snapshot of Le succès d'une réaction PCR dépend en grande partie de ... Calcul du ratio normalisé par un calibrateur.
 Principe de lamplification par PCR
Principe de lamplification par PCR
La PCR (Polymerase Chain Reaction ou réaction de polymérase en chaîne) est une technique Une méthode quantitative : la PCR en temps réel.
 Quoi faire avec des résultats de qPCR
Quoi faire avec des résultats de qPCR
Vous amplifiez une région de l'ARNm avec des oligos et une sonde fluorescente. La machine qPCR mesure l'intensité de fluorescence émise à chaque cycle. PCR.
 Document Cofrac SH GTA 04
Document Cofrac SH GTA 04
9.6 Vérification des critères de performances et méthodes de calcul . 9.6.1Vérification/validation d'une méthode quantitative.
 PCR quantitative en temps réel et PCR digitale
PCR quantitative en temps réel et PCR digitale
Principes de la PCR quantitative en temps réel : notion de Cq formats de fluorescence
 Évaluation de lacte de recherche ou de quantification du gène de
Évaluation de lacte de recherche ou de quantification du gène de
par une PCR quantitative (RT-QPCR). Actuellement seule la recherche du gène de fusion BCR-ABL est inscrite à la NABM. La re- cherche des transcrits BCR-ABL
 VÉRIFICATION ET VALIDATION DES MÉTHODES ANALYTIQUES
VÉRIFICATION ET VALIDATION DES MÉTHODES ANALYTIQUES
PR-GQ-011. Numéro d'approbation : Erreur ! Nom de propriété de document inconnu. Version 05. Page 4 de 14. Méthode quantitative : Elle fournit des résultats
 ÉVALUATION DE LA PERFORMANCE DU qPCR POUR LE
ÉVALUATION DE LA PERFORMANCE DU qPCR POUR LE
Mots clés : qPCR PCR quantitatif
 Comprendre des résultats de qPCR
Comprendre des résultats de qPCR
La machine qPCR mesure l'intensité de fluorescence émise à chaque cycle PCR. Pendant les premiers cycles il n'y a pas assez de fluorescence pour être
 Guide to Performing Relative Quantitation of Gene Expression
Guide to Performing Relative Quantitation of Gene Expression
Real-time quantitative PCR offers researchers a powerful tool for the quantitation of target nucleic acids To understand the value that real-time PCR provides over traditional PCR methods and to obtain basic information on chemistries and strategies you can review the following tutorials: Real Time PCR vs Traditional PCR
 Quantitative PCR Basics - Sigma-Aldrich
Quantitative PCR Basics - Sigma-Aldrich
Determination of PCR efficiency: Indirect methods • These use mathematical models to determine efficiency from individual amplification curves • Several methods available including: o second derivative maximum (Tichopad/Pfaffl) o mid-point slope determinations (Peirson) o linear regression using ‘window of linearity’ (Ramakers)
 qPCR Quantification Protocol Guide - Boston University
qPCR Quantification Protocol Guide - Boston University
quantifying DNA based on PCR qPCR tracks target concentration as a function of PCR cycle number in order to derive a quantitative estimate of the initial template concentration in a sample As with conventional PCR it uses a polymerase dNTPs and two primers designed to match sequences within a template
 Quantitative Polymerase Chain Reaction - Springer
Quantitative Polymerase Chain Reaction - Springer
Quantitative PCR 353 Although single-step RT-PCR protocols may be used including an initial RT step before the PCR is initiated we will focus here primarily on two-step protocols involving an initial cDNA synthesis followed by a separate PCR reaction
What is quantitative PCR?
Quantitative PCR (formally quantitative real-time PCR, qPCR) detection builds on the basic PCR technique and allows researchers to estimate the quantity of starting material in a sample.
What is the difference between qPCR and conventional PCR?
Since the products are detected as the reaction proceeds, qPCR has a much wider dynamic range of analysis than conventional, end-point PCR; from a single copy to around 1011copies are detectable within a single run.
What is the starting point of a real-time quantitative PCR assay?
Whatever the platform or chemistry involved, the starting point of a real-time assay is a tissue-specific RNA and the end point of a real-time reaction is an amplification plot.
Genomic Platform
Institute of research in immunology and cancer, University of Montreal www.genomique.iric.ca 1Quoi faire avec des résultats de qPCR
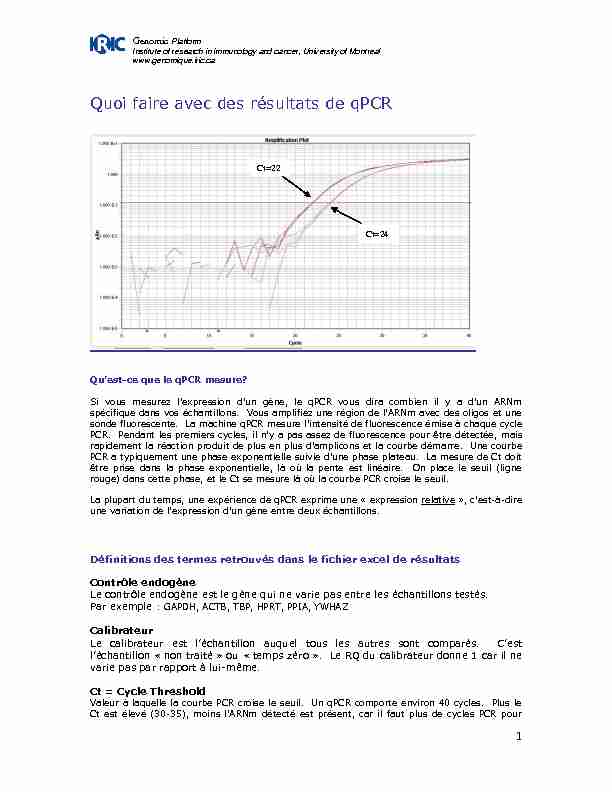
être prise dans la phase exponentielle, là où la pente est linéaire. On place le seuil (ligne
rouge) dans cette phase, et le Ct se mesure là où la courbe PCR croise le seuil. Définitions des termes retrouvés dans le fichier excel de résultatsContrôle endogène
Le contrôle endogène est le gène qui ne varie pas entre les échantillons testés.Par exemple : GAPDH, ACTB, TBP, HPRT, PPIA, YWHAZ
Calibrateur
varie pas par rapport à lui-même.Ct = Cycle Threshold
Valeur à laquelle la courbe PCR croise le seuil. Un qPCR comporte environ 40 cycles. Plus le Ct=22 Ct=24Genomic Platform
Institute of research in immunology and cancer, University of Montreal www.genomique.iric.ca 2fortement exprimé. Les contrôles endogènes ont souvent un Ct plus petit que les autres
gènes. Delta Ct = Ct gène ± Ct contrôle endogène Delta Delta Ct = ¨Ct échantillon1 ± ¨Ct calibrateurDelta Ct SD = Standard Deviation
Écart type calculé par le logiciel. Cette erreur reflète la qualité des triplicatas technique pour
le gène test et pour le gène endogène pour un même échantillon. Pour que la valeur soit
considérée comme valide, la SD doit se situer sous 0.25. Si la SD est au-dessus de 0.25, la valeur du RQ est alors considérée comme moins fiable.RQ = Relative quantification = 2-¨¨FP
échantillons ont une valeur par rapport à ce calibrateur. Nous considérons un résultat comme significatif lorsque le fold-change est de minimum deux, voulez observez un petit fold-change (1.5-2 fold), il vous faudra des échantillons parfaitementpropres, probablement faire des quadruplicata, penser à utiliser 2 essais pour le même gène,
et utiliser des contrôle positif avec des fold-change connus. de confiance place à 95%. Si vous voulez obtenir une vraie erreur biologique sur la valeur du RQ, vous aurez besoin de triplicata biologique. Ensuite, vous pouvez faire une moyenne des RQ ou des deltaCts destriplicatas. Si vous utilisez le 7900HT, le logiciel DataAssist peut le faire pour vous
(www.appliedbiosystems.com).Qualité générale des résultats
très prudent et assurez-vous que les répliquats sont beaux. Un DeltaCt SD<0.25 est bon. Cette erreur ne peut pas valider la valeur biologique valables statistiquement, il vous faut des répliquats biologiques. Si tous vos gènes sortent très tard (spécialement les contrôles endogènes), et queGenomic Platform
Institute of research in immunology and cancer, University of Montreal www.genomique.iric.ca 3Analyse avec plusieurs contrôles endogènes
La plupart du temps, un seul contrôle endogène est utilisé. Mais utiliser 2 ou 3
contrôles endogènes est de plus en plus recommandé, et améliorera la précision de vos résultats. Simplement testez plusieurs contrôles et sélectionnez les gènes qui varient le moins entre les échantillons (le logiciel Genorm peut être utilisé pour cette endogènes est possible directement avec le logiciel de base. Si vous utilisez le7900HT, télécharger le logiciel DataAssist (www.appliedbiosystems.com), et importer
une analyse " Individual Cts ». Ce logiciel peut aussi faire une analyse avec vos répliquats biologiques et faire un T test entre les groupes. les résultats.The MIQE guidelines
The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Bustin SA, Benes V, Garson JA, Hellemans J, Huggett J, Kubista M, Mueller R, Nolan T, Pfaffl MW, Shipley GL, Vandesompele J, Wittwer CT. Clin Chem. 2009 Apr;55(4):611-22. Epub 2009 Feb 26Genomic Platform
Institute of research in immunology and cancer, University of Montreal www.genomique.iric.ca 4Comment utiliser le logiciel SDS 2.2.2
Procurez-vous le logiciel SDS 2.2.2 disponible pour le téléchargement dans votre compte sur7900HT, le logiciel SDS 2.2.2 crée un fichier .sds avec les données brutes.
le logiciel).corrects. Vérifier si les contrôles endogènes sont bien identifiés dans la case " Task ».
Fermer le fichier.
3. Ouvrir un nouveau fichier : Relative Quantification (delta delta Ct) Study
4. En bas à droite, cliquer sur le bouton Add Plate et sélectionner le ou les fichier(s) .sds
correspondant(s).5. Cliquer sur la flèche verte.
courbes PCR.8. Pour visualiser un gène, sélectionner les puits correspondants, puis sélectionner le
9. Placer le Baseline en déplaçant la petite flèche rouge de droite en bas du graphique.
la première courbe commence.10. Déplacer le Seuil (ligne rouge) dans la section linéaire des courbes. Note : Le Baseline
11. Vérifier vos triplicata. Si vous voulez enlever un triplicata, sélectionner le puits,
faites attention quand vous faites des modifications car les sélections de puits sur différentes plaques se superposent. des Ct, cocher la case Individual ¨Cts.14. Dans le menu Fichier, sélectionner Export. Un fichier .txt sera produit.
Genomic Platform
Institute of research in immunology and cancer, University of Montreal www.genomique.iric.ca 515. Ouvrir le fichier exporté avec excel.
quotesdbs_dbs28.pdfusesText_34[PDF] calcul efficacité pcr quantitative
[PDF] 2 delta ct
[PDF] pcr quantitative relative
[PDF] delta delta ct calculation
[PDF] comment faire un transect
[PDF] comment réaliser un transect de végétation
[PDF] exemple de transect
[PDF] comment réaliser un transect végétal
[PDF] transect botanique
[PDF] transect definition
[PDF] protocole pcr taqman
[PDF] analyse résultats pcr quantitative
[PDF] pcr protocole pdf
[PDF] qpcr sybr green principe
